关于RNA甲基化修饰的研究成果在Nature,Science,Cell等高分期刊上频频亮相,并一次次刷新人们对生命科学的认知。拟南芥作为植物界中研究RNA甲基化修饰的先行者,许多学者将它作为研究对象,并与最新m6A、m5C RNA甲基化测序技术结合,证实到RNA甲基化广泛存在于拟南芥各个发育期,并揭示了RNA甲基化相关酶在特殊发育时期,如开花,叶片形成,种子发育,根部生长等过程中发挥重要作用。
小编全面整理了拟南芥RNA甲基化最新的研究成果,现从以下三方面进行阐述:
-
m6A RNA甲基化谱研究
-
m6A RNA甲基化酶分子机制研究
-
m5C RNA甲基化谱及甲基化酶分子机制研究
一、拟南芥m6A RNA甲基化谱研究汇总
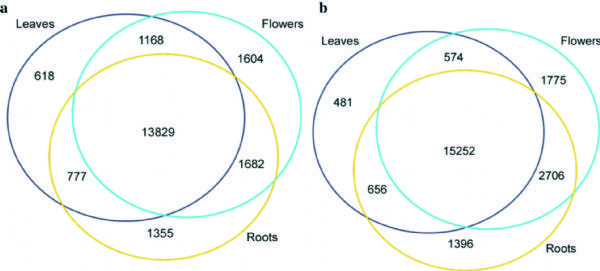
1. Genome Biology:拟南芥花叶根组织m6A RNA甲基化谱
影响因子:13.21
西北农林科技大学联合中科院和普渡大学,借助m6A RNA甲基化测序技术,对比拟南芥花,叶,根组织中(每种组织有两个生物学重复)m6ARNA甲基化情况。结果发现检测组织中m6A RNA甲基化修饰程度比人类高10%左右,占转录组的83%。
图1.比较拟南芥花,叶,根组织中m6A RNA甲基化情况
2. Nature Communications:不同品种拟南芥m6A RNA甲基化谱
影响因子:12.35
芝加哥大学联合北大,利用m6A RNA甲基化测序技术,对比Can-0和Hen-16品种拟南芥根组织中m6A RNA甲基化谱,发现m6A的分布在两个品种间高度保守。与人类m6A RNA甲基化位点分布情况相比,拟南芥m6A RNA甲基化位点在起始翻译区特异性高表达。
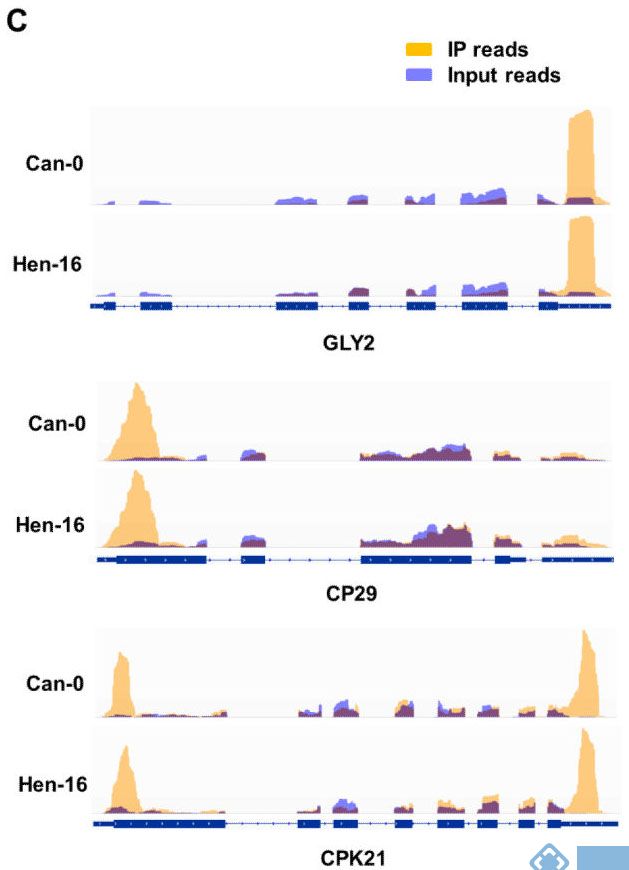
图2.不同品种,同一基因上甲基化peak有高度保守性
二、拟南芥m6A RNA甲基化酶研究汇总
m6A RNA甲基化修饰酶,主要包括Writer,Eraser和Reader,如METTL3/14/16,WTAP;ALKBH5等。那么,在植物中,RNA甲基化酶都研究到什么程度了?小编都给您整理好了,见下表。
|
拟南芥m6A RNA甲基化酶类型总结(截止到2018年6月) |
|
|
类别 |
基因 |
|
Writer |
MTA,MTB (分别与METTL3,METTL314同源) FIP37 (与WTAP同源) Virilizer(与KIAA1249同源) Hakai |
|
Eraser |
ALKBH10B |
|
Reader |
ECT2 |
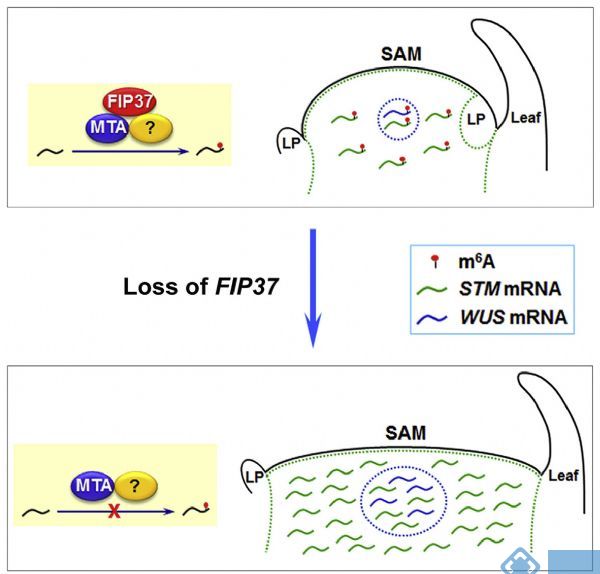
1.Developmental Cell:拟南芥m6A修饰调控芽尖细胞增殖
影响因子:11.91
由于拟南芥FIP37与人类甲基化转移酶WTAP高度同源,因此作者想看看FIP37的功能是否与人类的WTAP一样。带着这个问题,作者运用T-DNA突变法构建了FIP37的野生型和突变型,敲除后发现对茎尖分生组织增殖影响显著。又将正常,敲除或过表达FIP37拟南芥为样本,分别进行m6A RNA甲基化测序,结果发现敲除组m6A修饰水平显著低于正常和过表达组。通过RIP测序,证实FIP37能够直接与WUS和STM基因结合,进行m6A RNA甲基化修饰。
图3. m6A RNA甲基化调控FIP37影响拟南芥芽尖组织增殖
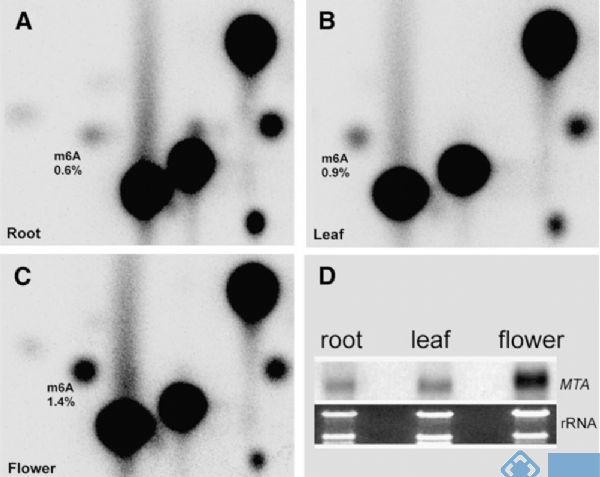
2.The Plant Cell:TLC法检测拟南芥m6A RNA甲基化修饰
影响因子:8.23
前篇文章的作者之所以挑FIP37来做机制研究,主要是缘于这篇文章的铺垫。在m6A RNA甲基化测序技术未成熟时期,先通过70年代就有应用的TLC法,检测到拟南芥花,叶,根组织中广泛存在m6A RNA甲基化修饰,在开花时期其甲基化修饰程度更高。作者接着应用酵母双杂交技术,证实了FIP37通过与MTA结合,形成甲基转移酶复合物。
图4. TLC法检测拟南芥组织中m6A RNA甲基化修饰
3.New Phytologist:拟南芥中存在多种m6A RNA甲基化转移酶
影响因子:7.43
马萨里克大学研究团队证实甲基化转移酶MTB和FIP37调控了m6A RNA甲基化修饰。分别敲降甲基化转移酶后,检测发现拟南芥根组织的增殖情况发生异常。另外,作者还检测到拟南芥的甲基化转移酶,Virilizer和Hakai。如果想研究植物RNA甲基化酶,“墙裂”推荐这篇作为敲门砖!
图5. TLC法检测敲降甲基化转移酶后m6A RNA甲基化修饰情况
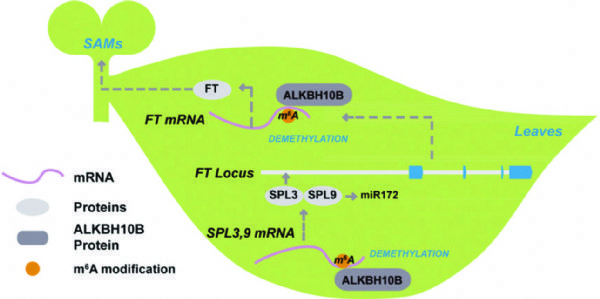
4.The Plant Cell:m6A RNA Eraser----ALKBH10调控拟南芥开花
影响因子:8.23
北大研究团队对拟南芥中去甲基化转移酶进行了研究,发现ALKBH10B在拟南芥开花期间高表达。敲降后,干扰组开花用时比对照组长。作者又通过m6A RNA甲基化测序,检测了敲降ALKBH10B样本后发现1000多个基因发生了差异的甲基化修饰。此文还找到几个与ALKBH10B直接作用的靶基因和miRNA,感兴趣的老师可以详细看看。
图6. ALKBH10B调控拟南芥开花的作用机制
接下来,小编将通过3篇文章介绍拟南芥与其m6A RNA甲基化Reader ECT2。有趣的是,这3篇文章发表在同一期刊:The Plant Cell;更有意思的是,它们见刊时间相差不到一个月。
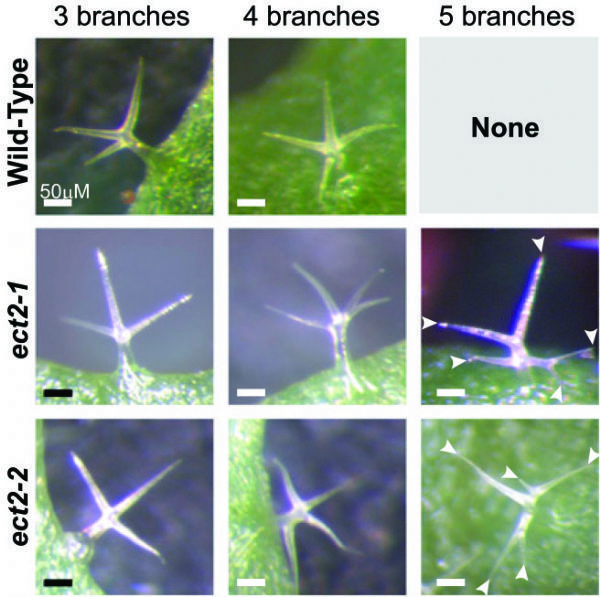
5.The Plant Cell:拟南芥m6A修饰调控ECT2的YTH结构,影响毛状分枝形态
影响因子:8.23
这篇文章的思路值得借鉴,作者首先以一般YTH结构域多在Reader上分布为依据,通过序列比对,发现拟南芥ECT2蛋白上存在许多YTH结构域,推测其能够识别m6A 结合位点。这个思路,不知云粉们是否觉得眼熟?之前,在m6A RNA甲基化---非编码RNA篇中就曾介绍过,发表在Nature上,作者在pri-miRNA上发现许多METTL3的motif,经过验证后发现这些motif的确与m6A RNA的甲基化转移酶有关。
图7. ECT2调控拟南芥毛状分枝的数量
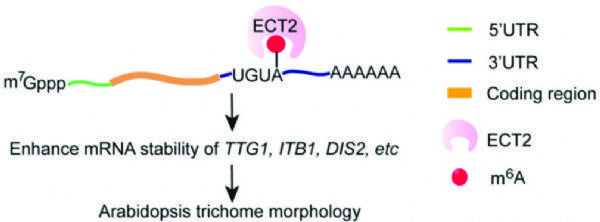
6.The Plant Cell:拟南芥ECT2的甲基化调控mRNA的稳定性并影响毛状分枝形态
影响因子:8.23
本篇文章重在筛选能与ECT2蛋白结合的靶基因,找到了3个,分别为TTG1、ITB1、DIS2。首先,作者根据自己的实验需求,开发了一套FA-CLIP(甲醛交联-免疫共沉淀)流程,成功找到发生甲基化RNA与ECT2结合位点,并确定结合区域为3'UTR。作者又应用凝胶迁移(EMSA)技术,证明3’UTR上的UGUA序列为植物特有,且可被ECT2特异性的识别。
图8. 拟南芥ECT2蛋白与靶基因结合的作用机制
7.The Plant Cell:YTH结构域调控m6A RNA甲基化修饰,影响叶片发育和形态
影响因子:8.23
哥本哈根大学研究团队对比研究了ECT2-4的结构,发现ECT2,3能通过与m6A结合位点结合,调控叶片生长,毛状体形态,而ECT4仅在ECT2,3都缺失时,才发挥作用。作者通过进化树和对已有转录组库的筛选,最终确定将ECT2-4作为重点来研究。通过定点突变m6A RNA甲基化位点,构建单敲/双敲/三敲的拟南芥模型及回补措施,分别对拟南芥叶片上的毛状分枝数量做统计,最终证实ECT2-4上的m6A RNA甲基化位点均具有调控作用。在拟南芥中,作者首次比对了人类与拟南芥ECT蛋白的结构,发现其N端包含许多无序的蛋白结构,此发现为推测Reader与靶基因结合从而形成聚合体提供理论依据。
图9.人类,酵母和拟南芥ECT1-4蛋白多重比对及甲基化位点分布
三、拟南芥m5C RNA甲基化谱及甲基化酶研究汇总
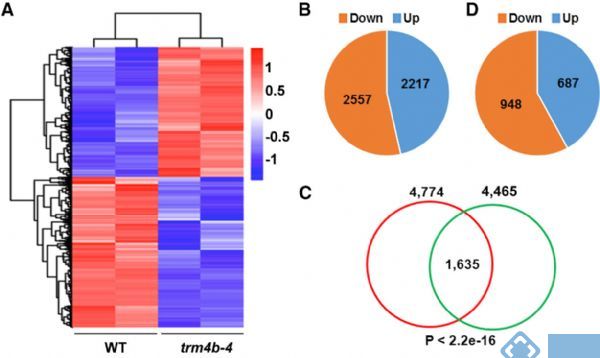
1. Molecular Plant:拟南芥m5C RNA甲基化谱及TRM4B酶功能
影响因子:9.33
中国农业科学院的谷晓峰课题组将RIP技术与传统转录组测序结合,检测到拟南芥中差异表达基因上平均有1-2个m5C甲基化位点。同时,证明了m5C甲基转移酶TRM4B及其突变体对拟南芥根发育的影响。
图10.RNA甲基化转录组测序检测基因差异表达模式
2. The Plant Cell:拟南芥m5C RNA甲基化谱及TRM4B酶对根的影响
影响因子:8.23
与上篇不同,本研究团队采用m5C RNA甲基化测序研究拟南芥中m5C甲基化谱,在种子,幼芽,根中发现了1000多个特异性的位点。敲低RNA m5C甲基转移酶TRM4B,造成tRNA稳定性的降低。研究人员还证实TRM4B突变体的初级根比野生型更短,同时对氧化的应激反应更敏感。
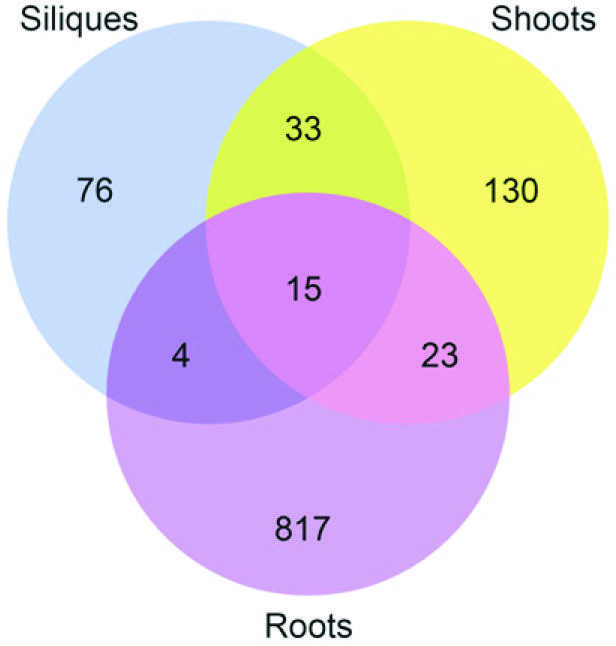
图11. m5C RNA甲基化测序检测组织间差异表达基因
云序相关产品推荐:
往期回顾
上海云序生物科技有限公司
Shanghai Cloud-seq Biotech Co.,Ltd
地址:上海市松江区莘砖公路518号20号楼3楼
电话:021-64878766
网站:www.cloud-seq.com.cn
邮箱:market@cloud-seq.com.cn