大多数的人类遗传病都是由点突变——DNA序列中单个的碱基错误所引起。然而,当前的基因组编辑方法不能够高效地校正细胞中的这些突变,而且经常导致随机的核苷酸插入或删除。
在一项新的研究中,来自美国哈佛大学的研究人员对CRISPR/Cas9技术进行改进,构建出一种新的“碱基编辑器(base editor)”,并且避免这些问题的发生。在人细胞系和小鼠细胞系中,这种碱基编辑器永久性地和高效地将碱基胞嘧啶(C)转化为碱基尿嘧啶(U),同时具有较低的编辑错误发生率。
在此之前,CRISPR/Cas9基因组编辑方法一直依赖于一种被称作同源重组修复(homology-directedrepair)的细胞机制,其中这种修复是由基因组DNA上的双链断裂触发的。科学家们给细胞导入一种含有目标序列的DNA模板,并利用酶Cas9在这种细胞的基因组靶位点上进行切割,导致基因组发生双链断裂,然后等待一段时候后观察这种同源重组修复是否将这种模板整合到基因组中,将双链重新连接在一起。不幸的是,这种方法是低效率的(整合很少发生),而且经常在断裂点附近以随机indel的形式引入新的碱基错误,从而使得它不适合用于点突变的治疗性地校正。
因此,在哈佛大学化学家和化学生物学家David Liu的领导下,研究人员尝试了一种不同的方法。首先,他们让Cas9部分失活,使得它不能够在细胞基因组上产生双链断裂。他们然后让Cas9与一种被称作胞苷脱氨酶的酶偶联在一起,这样就可在不用切割DNA的情形下,直接催化C变成U(本质上等同于胸腺嘧啶T)。将这种偶联物导入细胞中,会在细胞基因组靶位点上产生一对错配的碱基对:一条链上新产生的U与另一条链上初始碱基G错配。
与此同时,还有两篇文章同一天出现在线Nature期刊上。这两篇文章都涉及Cas9的一种潜在替代者:Cpf1酶。CRISPR/Cpf1在细胞基因组靶位点上进行切割,产生粘性末端(sticky end)---切割的DNA上存在的突出末端,在DNA双链断裂位点的两边留下未配对的碱基---而不是利用Cas9切割DNA双链产生的平头末端(blunt end)。
在其中的一篇文章中,来自德国马克斯普朗克感染生物学研究所的Emmanuelle Charpentier和同事们证实不同于Cas9,Cpf1除了切割DNA外,还对RNA进行加工。
在另外一篇文章中,来自中国哈尔滨工业大学生命科学与技术学院的黄志伟(Zhiwei Huang)教授和同事们解析出CRISPR/Cpf1的晶体结构。
本文引自生物通
参考文献
1.D.Dong et al. (2016) The crystal structure of Cpf1 in complex with CRISPR RNA. Nature.doi:10.1038/nature17944.
2.I.Fonfara et al. (2016) The CRISPR-associated DNA-cleaving enzyme Cpf1 also processesprecursor CRISPR RNA. Nature. doi:10.1038/nature17945.
3.A.C.Komor et al. (2016) Programmable editing of a target base in genomic DNA withoutdouble-stranded DNA cleavage. Nature. doi:10.1038/nature17946.
延伸阅读
OligoMix®成功扮演CRISPR的Guide RNAs

在CRISPR-Cas9突变筛选中,研究人员需要将能够靶向成千上万个基因位点的guide RNAs克隆到病毒载体中,然后“投递”到靶向细胞中。通过鉴定guide RNAs在设定表型细胞中的富集情况,研究人员能够系统并快速地鉴定与特定表型相关的基因。为了使大规模筛选试验成为可能,就需要大规模合成guide RNAs。
OligoMix®正为那些希望大规模合成guide RNAs文库的研究人员提供了一套独一无二的解决方案。研究人员可以快速合成包含数千条完全定制化的序列文库,这些特异性单链寡核苷酸序列可识别基因组中特定的区域。
下面举几个OligoMix®作为CRISPR Guide RNAs的成功应用案例
Nat Biotechnol (2016) High throughput mapping of regulatory DNA.(IF=41.514)
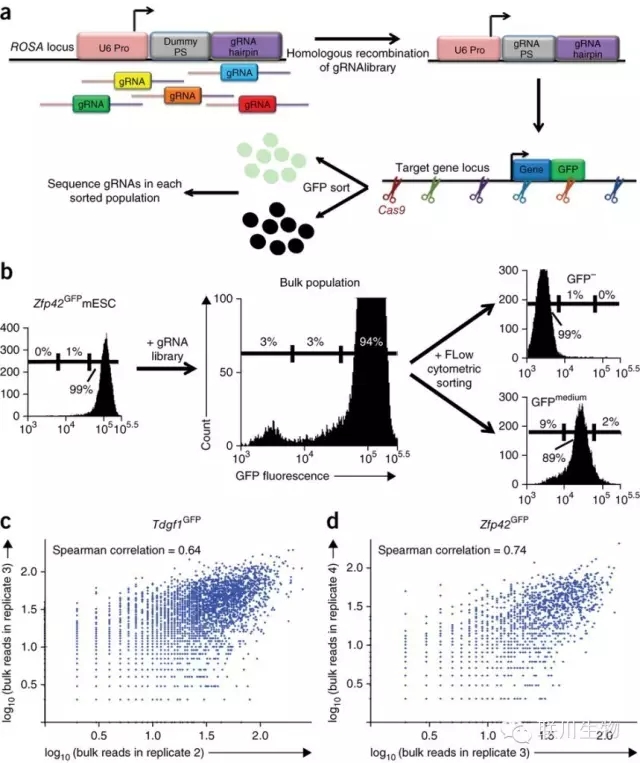
图1 MERA实验方案
来自MIT(Gifford实验室)和Brigham and Women’s Hospital的研究人员证实,OligoMix®合成的guide RNAs文库在MERA技术中具备有效性。MERA技术全称是Multiplexed Editing Regulatory Assay,即基于CRISPR-Cas9的多重基因编辑调控实验的筛选方法。
Genome Res (2016) A new class of temporarily phenotypic enhancers identified by CRISPR/Cas9-mediated genetic screening.(IF=14.630)
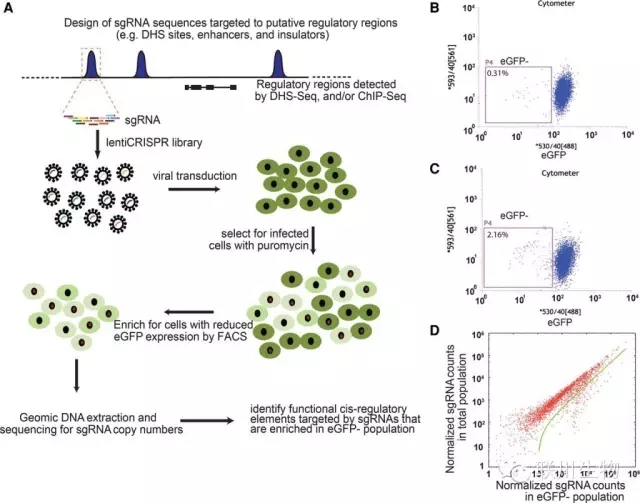
图2 鉴定顺式调控元件的高通量CRISPR/Cas9介导筛选方案
Ludwig Institute for Cancer Research任兵教授团队以Oligomix®作为Guide RNAs,成功开发出一种基于CRISPR/Cas9基因组编辑的高通量筛选技术,可用于功能性筛选人类基因组中的顺式调控元件。研究团队将这一策略用于人类胚胎干细胞中POU5F1基因上,不仅揭示出一些经典的顺式调控元件,还发现一类非常规的调控元件。这些非常规的调控元件能够以一种意想不到的方式进行转录调控。
Methods Enzymol (2014) Adapting CRISPR/Cas9 for functional genomics screens.
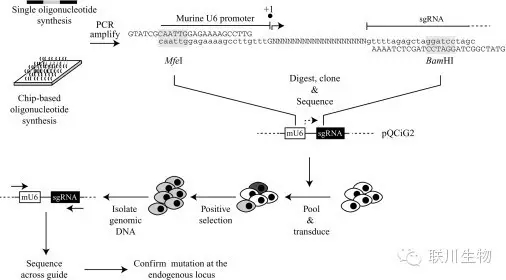
图3 sgRNA文库合成与混合筛选策略
McGill University研究人员证实将寡核苷酸库(Oligomix®)克隆到逆转录病毒表达载体,可同时传递Cas9核酸酶和sgRNAs。这种方法为高通量功能基因组筛选提供了稳定和可重复性表达的基因编辑工具。
回复“crispr”,查看做crispr的神器。


